Technologischer Fortschritt
Aufgrund rapider Fortschritte in der Entwicklung hochmoderner Technologien und Informatik-Tools, welche ursprünglich der Generierung und Verarbeitung biologischer Datensätze in der Biomedizin dienten, werden die neuen Methoden inzwischen auch in anderen Wirtschaftsbereichen erfolgreich eingesetzt. Omik-Technologien ermöglichen eine ganzheitliche Sicht auf die molekularen Charakteristika und Abläufe einer Zelle, eines Gewebes oder eines kompletten Organismus. „Omik“ bezeichnet daher neuartige Gesamtkonzepte zur Analyse des kompletten genetischen oder molekularen Profils von Organismen mit Hilfe integrativer Pipelines, die aufzeigen wie komplexe Interaktionen zwischen Genen und Molekülen den Phänotyp beeinflussen, beispielsweise die Krankheitssymptome eines Patienten. Umfassende technologische Verbesserungen bezüglich Next-Generation-Sequencing, Microarray, Massenspektrometrie und Nuklearmagnetresonanz haben die biologische und biomedizinische Forschung der letzten Jahre grundlegend verändert. Die Analyse von Omik-Daten ist ein expandierendes Fachgebiet und bietet durch die Erschaffung immer neuer statistischer Methoden nicht nur dem medizinischen Bereich neue Perspektiven. [1,2,3,4,5] Beispiele an verschiedenen Omik-Datentypen: [2,3,4,5]
- Genomik: Gesamtheit aller Gene eines Organismus
- Proteomik: Gesamtheit aller Proteine, die in einem Organismus produziert werden
- Transkriptomik: Gesamtheit aller RNA-Moleküle
- Pharmakogenomik: Der Einfluss genetischer Veränderungen auf die Wirkung von Medikamenten
Verglichen mit einzelnen Omik-Analysen bieten Multi-Omik-Abfragen einen umfangreicheren Informationsgewinn für Forscher, von der ursprünglichen Krankheitsursache (genetisch, umwelt- oder entwicklungsbedingt) bis zu funktionalen Auswirkungen oder relevanten Interaktionen. [2,3]
Computergestützte Biologie
Die Bioinformatik nutzt computergestützte Tools zur Verwaltung und Analyse biologischer Daten aus großen Datensätzen. Der integrative Ansatz, solche Tools mit entsprechenden Datenbanken zu kombinieren, wird Computergestützte Biologie genannt und erlaubt die Erschaffung ganzer Karten mit zellulären und physiologischen Signalwegen. Methoden der Bioinformatik ermöglichen die ganzheitliche Darstellung einer Zelle bzw. eines Organismus sowie Vorhersagen zu hochkomplexen Systemen wie Phänotypen oder Netzwerken zellulärer Interaktionen. (Abb. 1). [6] Die Zusammenführung zwischen Omik und Bioinformatik stellt die Basis der Systembiologie dar. Dieses Studiengebiet erzeugt Modellorganismen, um ein tieferes Verständnis komplexer biologischer Interaktionen innerhalb von Zellen oder Geweben auf Gen-, Protein- oder Metabolit-Ebene zu erlangen. [2,5] Aber da die Generierung solcher Datensätze immer weniger Zeit und Kosten beansprucht, bietet die Integrierung von Omik-Daten sowohl bahnbrechende Möglichkeiten als auch immense Herausforderungen für Biologen, Biostatistiker und Biomathematiker.
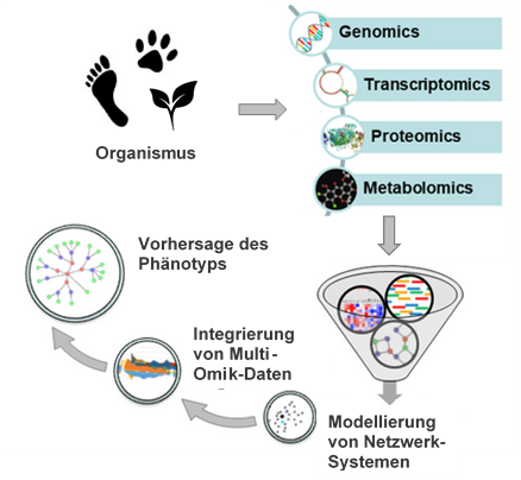
Herausforderungen
Heutzutage gibt es unzählige web-basierte Lösungen für die Speicherung, Analyse und den Austausch von Daten. Beispiele allgemein bekannter Portale mit Download-Möglichkeit von Omik-Daten sind Gene Expression Omnibus, ArrayExpress, Expression Atlas oder Ensembl. Andere Portale bieten zusätzlich Optionen zur Analyse der entsprechenden Daten in interaktiven und vielschichten Ebenen, wie zum Beispiel NCBI, UCSC oder der Human Brain Atlas. Aufgrund der stetig zunehmenden Zahl bioinformatischer Tools wurden zudem spezielle Plattformen zur Auflistung der Tools geschaffen, um deren effektiven Nutzen zu gewährleisten. EBI beispielweise stellt ein umfangreiches Portal mit einer Vielzahl nach Themen sortierter Datenbanken und Tools zur Verfügung, wohingegen OMICtools eine Bibliothek mit speziellen Softwarelösungen, Datenbanken und Plattformen zur Verarbeitung und Analyse von großen Datenmengen bietet. [4,5] Aber trotz der Existenz zahlloser Omik-Tools zur Analyse ganzer Netzwerke, Darstellung von Signalwegen, Genomalignments, Visualisierung und vieler anderer Einsatzmöglichkeiten kann kaum eine dieser Lösungen 3 oder mehr verschiedene Omik-Datensätze integrieren. Die Verwaltung und Integrierung multidimensionaler Daten bleibt somit weiterhin schwierig, auch weil jede Omik-Analyse täglich Dateien in Tera- oder Peta-Byte-Größe erzeugen kann. Aktuell bestehen die größten Herausforderungen in den biologischen und technischen Unterschieden bezüglich der Aufbereitung, Speicherung und Verarbeitung von Daten, der wissenschaftlichen Kontexterstellung, der statistischen Validierung sowie der Rechenleistung. Außerdem mangelt es an stabilen Pipelines, die zusätzliche Datentypen integrieren können. Innovative Ansätze zur Einbindung von Omik-Daten werden gerade im Bereich der Ernährungswissenschaften, Mikrobiomanalysen, Genotyp-Phänotyp-Interaktion, Systembiologie und der Krankheitsforschung dringend benötigt. [3,4,5]
Bedeutung der Omik Tools in der Medizin
Umfangreiches Pofiling ermöglicht mittlerweile tiefe Einblicke in die Entstehung schwerer und chronischer Krankheiten wie Krebs, Herz- oder Infektionskrankheiten, erleichtert die Suche nach diagnostischen Markern oder effizienten neuartigen Therapien und erlaubt die Vorhersage von Therapieerfolgen. Die Identifizierung prädiktiver und frühdiagnostischer Marker ist gerade in der Krebstherapie von großem Vorteil, da einige Krebsarten erst in fortgeschrittenem Stadium diagnostiziert werden und die Überlebenschancen demnach nur gering sind. [1,5] Integrative Multi-Omik-Analysen sollen hierbei eine ganzheitliche Sicht auf Krankheitsmechanismen gewähren, die die Ausführung normaler Zellfunktionen stören und zu einem Fortschreiten der Krankheit und zu Arzneimittelresistenz führen. Doch während computergestützte Analysen einzelner Omik-Datensätze inzwischen gut etabliert sind, sind Ansätze zur Integrierung von Multi-Omik-Daten noch weit von der standardmäßigen Anwendung entfernt. Um mit der enormen Geschwindigkeit der Datengenerierung und des Wachstums von biologischem Wissen mithalten zu können müssen existierende Methoden erweitert oder generalisiert werden. Neue Tools müssen zudem die Komplexität und die vielschichtigen Ebenen der verfügbaren Informationen bewältigen können. [7]
Bedeutung der Omik Tools in anderen Sektoren
Nicht nur der medizinische Bereich profitiert von spezialisierten Bioinformatik-Tools, sondern auch andere wichtige Wirtschaftssektoren wie die Landwirtschaft oder die Pflanzen- und Tierwissenschaft. Honigbienen beispielweise sind als Bestäuber unersetzbar für die globale Landwirtschaft. Der Gesundheitszustand der Honigbienen hat sich in den letzten Jahrzehnten rapide verschlechtert, wobei Bienenzüchter seit 2007 teilweise mehr als ein Viertel ihrer Kolonien pro Jahr verlieren. Die Gründe für das Bienensterben sind komplex, zeit- und ortsunabhängig und sehr schwierig zu definieren. Das kanadische Projekt BeeCSI, welches 2018 gestartet wurde, will nun mit Hilfe genomischer Tools eine neue Plattform zur Einschätzung und Diagnose der momentanen Bienengesundheit entwickeln, die auf Stressfaktoren und entsprechenden Biomarkern basiert. Zwei Spezialisten der York University und der University of British Columbia leiten ein nationales Team aus Forschern, Bioinformatikern, Bienenzüchtern und diagnostischen Laboren. BeeCSI soll in Kanada eine industrielle Modernisierung in Form dieses neuen Tools herbeiführen, welches eine schnelle Einschätzung des Gesundheitszustands von Bienenkolonien erlaubt und somit den Bienenzüchtern ermöglicht, frühzeitig entsprechende Maßnahmen gegen das Bienensterben zu ergreifen. Das 10 Mio $ Projekt wird von Genome Canada und Ontario Genomics gefördert. [8]
Perspektiven
Studien in Genomik, Transkriptomik und Proteomik haben unser Verständnis der zellulären Komplexität und Heterogenität maßgeblich beeinflusst. Aufgrund stetig fallender Kosten der Omik-Analysen können im medizinischen Bereich inzwischen noch mehr Datentypen zur Erstellung individueller Behandlungspläne integriert werden. Jedoch ist der Weg der Standardisierung langwierig: Die Generierung skalenübergreifender Omik-Daten, die Entwicklung neuer analytischer Methoden, eine Anpassung dieser Methoden auf spezifische Krankheiten sowie die Wiederholung dieser Prozesse für weitere Krankheiten sind fundamentale Aufgaben in der Omik-Forschung, können jedoch von einem Team alleine nicht bewältigt werden. Um den aktiven Informationsfluss zwischen verschiedenen Fachgebieten zu gewährleisten müssen koordinierte Maßnahmen vieler fachlich geeigneter Teams getroffen werden, was schließlich eine Standardisierung von Datenformaten und Pipelines für integrierte Multi-Omik-Analysen ermöglichen kann. [1,2,4,5] Ansprechpartner: Kerstin Hammer Quellen




