 CRISPR/Cas ist zurzeit in aller Munde. Deswegen gibt es diese Woche einen Beitrag über DAS Genome-Editing-Werkzeug und aufgrund des Welt-AIDS-Tages letzten Samstag erste Erfolge in der HIV-Behandlung und einer Nachricht, die letzten Monat viele schockiert hat.
CRISPR/Cas ist zurzeit in aller Munde. Deswegen gibt es diese Woche einen Beitrag über DAS Genome-Editing-Werkzeug und aufgrund des Welt-AIDS-Tages letzten Samstag erste Erfolge in der HIV-Behandlung und einer Nachricht, die letzten Monat viele schockiert hat.
Die DNS
 Im Black-Friday-Beitrag ging es schon grob um Chromosomen und DNS. Kurz zur Erinnerung: Die DNS ist ein langes, kettenförmiges Polymer, das aus einzelnen Bausteinen, den Nukleotiden besteht. Ein Nukleotid setzt sich zusammen aus einem Phosphat, einem Zucker und aus einer von den vier Basen (Adenin (A), Cytosin (C), Guanin (G) und Thymin (T)). Watson & Crick entschlüsselten 1953 die Struktur:1 sie stellten fest, dass die DNS eine Doppelhelix bildet. Zwei solcher kettenförmigen Polymere, auch Einzelstränge genannt, lagern sich in entgegengesetzter Richtung zusammen, was sich komplementär nennt. Das Rückgrat bildet hierbei das Phosphat und der Zucker, während sich die Basen über Wasserstoffbrückenbindungen miteinander paaren und zwar immer die Base Adenin mit Thymin, und Cytosin mit Guanin.
Im Black-Friday-Beitrag ging es schon grob um Chromosomen und DNS. Kurz zur Erinnerung: Die DNS ist ein langes, kettenförmiges Polymer, das aus einzelnen Bausteinen, den Nukleotiden besteht. Ein Nukleotid setzt sich zusammen aus einem Phosphat, einem Zucker und aus einer von den vier Basen (Adenin (A), Cytosin (C), Guanin (G) und Thymin (T)). Watson & Crick entschlüsselten 1953 die Struktur:1 sie stellten fest, dass die DNS eine Doppelhelix bildet. Zwei solcher kettenförmigen Polymere, auch Einzelstränge genannt, lagern sich in entgegengesetzter Richtung zusammen, was sich komplementär nennt. Das Rückgrat bildet hierbei das Phosphat und der Zucker, während sich die Basen über Wasserstoffbrückenbindungen miteinander paaren und zwar immer die Base Adenin mit Thymin, und Cytosin mit Guanin.
CRISPR/Cas – Definition
Um nun zu CRISPR zu kommen: CRISPR steht für clustered regularly interspaced short palindromic repeats, auf Deutsch also gruppierte, kurze, palindromische Wiederholungen mit regelmäßigen Abständen. Ein Palindrom ist in diesem Fall eine Sequenz, bei der die Basen auf dem einen DNS-Strang die gleiche Reihenfolge aufweisen, wie die auf dem komplementären Strang – nur in entgegen gesetzter Richtung. Sie weisen also gegenläufig die gleiche Sequenz auf. 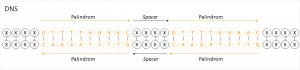 Auch wenn solchen Sequenzen ein Promotor vorangeschaltet ist, der dafür zuständig ist, dass die DNS zuerst in RNS (= Ribonukleinsäure) transkribiert und dann in Proteine translatiert wird, ergeben diese jedoch kein funktionstüchtiges Protein. Lange Zeit wurde der Sinn dahinter nicht verstanden.
Auch wenn solchen Sequenzen ein Promotor vorangeschaltet ist, der dafür zuständig ist, dass die DNS zuerst in RNS (= Ribonukleinsäure) transkribiert und dann in Proteine translatiert wird, ergeben diese jedoch kein funktionstüchtiges Protein. Lange Zeit wurde der Sinn dahinter nicht verstanden.
CRISPR/Cas – Geschichte
Bereits 1987 entdeckte Yoshizumi Ishino in dem Bakterienstamm Escherichia coli K12 die Existenz sich wiederholender DNS-Abschnitte. Er identifizierte gleiche Sequenzen aus jeweils 29 Nukleotiden, die von variablen Regionen aus 32 Nukleotiden unterbrochen wurden.2 1993 wurde ein ähnliches Motiv in der DNS von Mycobacterium tuberculosis entdeckt, einem der Überträger der Tuberkulose.3 2002 wurden ähnliche Strukturen im Genom vieler Prokaryonten, zu denen Bakterien und Archaeen zählen, beobachtet und der Begriff CRISPR geprägt. Zusätzlich wurde eine Gruppe von Genen entdeckt, die in allen untersuchten Organismen nahe der CRISPR-Sequenz liegen und wurden daher als CRISPR-associated genes (Cas-Gene) bezeichnet.4 2005 wurde heraus gefunden, dass die Sequenzen, die zwischen den Palindrom-Sequenzen liegen und als Spacer (Platzhalter) bezeichnet werden, mit der DNS von Viren identisch ist.5,6,7  Schätzungen zufolge gibt es ca. 4-6×1030 Prokaryonten auf der Erde und etwa zehnmal so viele Viren. Da diese, wie im letzten Beitrag bereits erwähnt, zum Überleben auf Wirtszellen angewiesen sind, vernichten sie alle zwei Tage ca. die Hälfte von ihnen. Es wurde somit die Hypothese aufgestellt, dass CRISPR-Bakterien und -Archaeen gegen diese Fremd-DNS der Viren verteidigt und somit als Immunsystem fungiert.5,6,7 2007 wurde diese Behauptung durch Barrangov und Kollegen bestätigt, die gezeigt haben, dass Bakterien, die mit Phagen (Gruppen von Viren) infiziert wurden, Teile deren DNS als Spacer zwischen die CRISPR-Bereiche integriert haben und somit eine Immunität gegenüber diesen Erregern aufgebaut haben: künstlich eingefügte Spacer, die die Sequenz von Viren enthielten, haben die Bakterien gegenüber der Fremd-DNS resistent gemacht. Das Experiment funktionierte auch umgekehrt – die Bakterien verloren diese erworbene Resistenz wieder, wenn die Sequenzen deletiert wurden.8 Somit wurde gezeigt, dass Prokaryonten ein adaptives Immunsystem besitzen, mit dem sie sich DNS-Sequenzen von Erregern merken, ähnlich beim Menschen durch Antikörper, und bei erneuter Infektion deren DNS zerschneiden können. Im Detail funktioniert der Mechanismus wohl so: bei einer Infektion wird die DNS der Viren in kleine Fragmente aufgespalten und in die CRISPR-Abschnitte eingefügt. Bei einem erneuten Befall werden diese Abschnitte in RNS umgeschrieben – deswegen auch der vorangeschaltete Promotor – und die virale DNS überprüft. Stimmt diese mit der „gespeicherten“ überein, wird sie durch DNS-schneidende Proteine, den Cas-Proteinen, zerstückelt und dadurch zerstört.9
Schätzungen zufolge gibt es ca. 4-6×1030 Prokaryonten auf der Erde und etwa zehnmal so viele Viren. Da diese, wie im letzten Beitrag bereits erwähnt, zum Überleben auf Wirtszellen angewiesen sind, vernichten sie alle zwei Tage ca. die Hälfte von ihnen. Es wurde somit die Hypothese aufgestellt, dass CRISPR-Bakterien und -Archaeen gegen diese Fremd-DNS der Viren verteidigt und somit als Immunsystem fungiert.5,6,7 2007 wurde diese Behauptung durch Barrangov und Kollegen bestätigt, die gezeigt haben, dass Bakterien, die mit Phagen (Gruppen von Viren) infiziert wurden, Teile deren DNS als Spacer zwischen die CRISPR-Bereiche integriert haben und somit eine Immunität gegenüber diesen Erregern aufgebaut haben: künstlich eingefügte Spacer, die die Sequenz von Viren enthielten, haben die Bakterien gegenüber der Fremd-DNS resistent gemacht. Das Experiment funktionierte auch umgekehrt – die Bakterien verloren diese erworbene Resistenz wieder, wenn die Sequenzen deletiert wurden.8 Somit wurde gezeigt, dass Prokaryonten ein adaptives Immunsystem besitzen, mit dem sie sich DNS-Sequenzen von Erregern merken, ähnlich beim Menschen durch Antikörper, und bei erneuter Infektion deren DNS zerschneiden können. Im Detail funktioniert der Mechanismus wohl so: bei einer Infektion wird die DNS der Viren in kleine Fragmente aufgespalten und in die CRISPR-Abschnitte eingefügt. Bei einem erneuten Befall werden diese Abschnitte in RNS umgeschrieben – deswegen auch der vorangeschaltete Promotor – und die virale DNS überprüft. Stimmt diese mit der „gespeicherten“ überein, wird sie durch DNS-schneidende Proteine, den Cas-Proteinen, zerstückelt und dadurch zerstört.9
Genome-Editing
 Dieses Erkennen und Schneiden einer Sequenz wird schon länger beim sogenannten Genome-Editing genutzt. Genome-Editing ist ein präzises Instrument, mit dem die DNS gezielt verändern wird. Gene können damit in eine bereits vorhandene Sequenz eingefügt, entfernt oder komplett ausgetauscht werden. Auch einzelne Nukleotide können somit geändert werden. Eine neue Ära in der Molekularbiologie stellte die Entdeckung von Restriktionsenzymen in den 1970er Jahren dar: diese Enzyme erkennen charakteristische Sequenzen und schneiden diese.10 Das Problem hierbei ist, dass sich der Zielort schwer vorhersagen lässt, da die Erkennungssequenz meist nur wenige Basenpaare lang ist und somit vermehrt im Genom vorkommt.
Dieses Erkennen und Schneiden einer Sequenz wird schon länger beim sogenannten Genome-Editing genutzt. Genome-Editing ist ein präzises Instrument, mit dem die DNS gezielt verändern wird. Gene können damit in eine bereits vorhandene Sequenz eingefügt, entfernt oder komplett ausgetauscht werden. Auch einzelne Nukleotide können somit geändert werden. Eine neue Ära in der Molekularbiologie stellte die Entdeckung von Restriktionsenzymen in den 1970er Jahren dar: diese Enzyme erkennen charakteristische Sequenzen und schneiden diese.10 Das Problem hierbei ist, dass sich der Zielort schwer vorhersagen lässt, da die Erkennungssequenz meist nur wenige Basenpaare lang ist und somit vermehrt im Genom vorkommt. 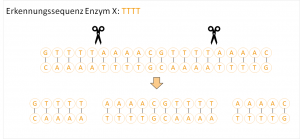 Mit CRISPR/Cas werden solche ungewollten Effekte weitgehend verhindert. Zusätzlich ist dieses Verfahren zuverlässig, schnell und kostengünstig. Das Funktionsprinzip beruht auch wie bei andere Genome-Editing Methoden auf folgenden drei Schritten: Es muss zuerst die Stelle im Genom gefunden werden, die gezielt verändert werden soll. Es wird eine „Sonde“ bzw. ein Vektor konstruiert, der die gewünschte Zielsequenz, auch als gRNS (guideRNS) bezeichnet, und die Sequenz für die molekular Schere – dem Protein Cas9 – enthält. Am Zielort angekommen, schneidet Cas9 den DNS-Doppelstrang genau an der gewünschten Sequenz. Zum Schluss kommt das zelleigene Reparatursystem ins Spiel und fügt die durchtrennten DNS-Stränge wieder zusammen.11 Hier hat die Zelle zwei Möglichkeiten: Nicht-homologe Endverknüpfung (Non-homologous end joining NHEJ) oder homologe Rekombination (Homology directed repair, HDR).
Mit CRISPR/Cas werden solche ungewollten Effekte weitgehend verhindert. Zusätzlich ist dieses Verfahren zuverlässig, schnell und kostengünstig. Das Funktionsprinzip beruht auch wie bei andere Genome-Editing Methoden auf folgenden drei Schritten: Es muss zuerst die Stelle im Genom gefunden werden, die gezielt verändert werden soll. Es wird eine „Sonde“ bzw. ein Vektor konstruiert, der die gewünschte Zielsequenz, auch als gRNS (guideRNS) bezeichnet, und die Sequenz für die molekular Schere – dem Protein Cas9 – enthält. Am Zielort angekommen, schneidet Cas9 den DNS-Doppelstrang genau an der gewünschten Sequenz. Zum Schluss kommt das zelleigene Reparatursystem ins Spiel und fügt die durchtrennten DNS-Stränge wieder zusammen.11 Hier hat die Zelle zwei Möglichkeiten: Nicht-homologe Endverknüpfung (Non-homologous end joining NHEJ) oder homologe Rekombination (Homology directed repair, HDR). 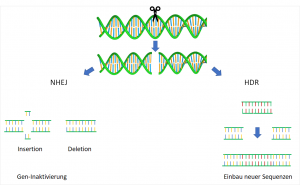 Bei NHEJ entstehen zufällige Insertionen, bzw. Deletionen durch einzelne Nukleotide, die zu einer Gen-Inaktivierung führen, da der Leserahmen (ORF; der Bereich, der für ein potentielles Protein codiert) dadurch verschoben wird. Bei HDR wird ein Template (Vorlage) hinzugefügt, um der Zelle genau vorzugeben, welche neue Sequenzen eingebaut werden sollen.
Bei NHEJ entstehen zufällige Insertionen, bzw. Deletionen durch einzelne Nukleotide, die zu einer Gen-Inaktivierung führen, da der Leserahmen (ORF; der Bereich, der für ein potentielles Protein codiert) dadurch verschoben wird. Bei HDR wird ein Template (Vorlage) hinzugefügt, um der Zelle genau vorzugeben, welche neue Sequenzen eingebaut werden sollen.
Gentherapie
 Bisher wurden schon Patienten mit defekten Genen durch eine klassischen Gentherapie behandelt. Hierbei wurden sie mit einem Virus behandelt, der das neue funktionsfähige Gen trägt und in die betroffenen Zellen einbaut. Doch auch hier ist das Problem, dass es sich nicht vorhersagen lässt, wo genau im Genom das entsprechende Gen eingebaut wird.12 Mit CRISPR wird dieser Umstand umgangen und der Zielort genau vorherbestimmt.
Bisher wurden schon Patienten mit defekten Genen durch eine klassischen Gentherapie behandelt. Hierbei wurden sie mit einem Virus behandelt, der das neue funktionsfähige Gen trägt und in die betroffenen Zellen einbaut. Doch auch hier ist das Problem, dass es sich nicht vorhersagen lässt, wo genau im Genom das entsprechende Gen eingebaut wird.12 Mit CRISPR wird dieser Umstand umgangen und der Zielort genau vorherbestimmt.
CRISPR/Cas in der HIV-Therapie
Es wurden schon erste Erfolge in der Behandlung mittels CRISPR/Cas verzeichnet, beispielsweise in der HIV-Therapie. Eine Infektion mit dem HI-Virus ist mittels retrovirale Therapie (ART) weitgehend unter Kontrolle gebracht, eine komplette Heilung gibt es jedoch noch nicht. Versuche haben gezeigt, dass CRISPR/Cas effektiv HI-Viren bekämpft. Mithilfe der molekularen Schere hat das Team um Kamel Khalili und Won-Bin Young sowohl in Mäusen, als auch in menschlichen Immunzellen, die auf „humanisierte“ Mäuse übertrugen wurden, das virale Erbgut aus mehr als 95% der infizierten Zellen entfernt.13  Eine Aussicht auf eine Anwendung direkt am Menschen ist jedoch noch nicht in Sicht. Denn auch diese Methode ist nicht ohne Risiken. Zum einen werden nur 95% entfernt, der Rest reicht, um die Krankheit erneut ausbrechen zu lassen. Zum anderen wird für den Transport der CRISPR-DNS ein Virus verwendet, der im Körper eine Immunantwort auslöst und somit nur einmal eingesetzt werden kann. Außerdem stehen die Transportviren in Verdacht, Tumore auszulösen.14 Eine weitere Arbeitsgruppe hat dieses Jahr mittels CRISPR/Cas die regulatorischen Gene von HIV zerstört, sodass dieser nicht weiter in der Lage war, sich in den infizierten Zellen zu vermehren. Dafür haben sie einen Vektor konstruiert, der sowohl Cas9 als auch eine von sechs verschiedenen gRNSs codiert. Diese Vektoren wurden in Zellen transfiziert, die die regulatorischen Proteine des Virus (Tat und Rev) exprimieren. Dadurch wurde eine signifikante Erniedrigung in der Expression und Funktion der Proteine verzeichnet, ohne dass dies das Überleben der Zellen beeinträchtigt hat.15 Durch diese beiden Veröffentlichungen zeigt sich, dass es durch CRISPR eine Heilung für HIV-infizierte Menschen geben könnte.
Eine Aussicht auf eine Anwendung direkt am Menschen ist jedoch noch nicht in Sicht. Denn auch diese Methode ist nicht ohne Risiken. Zum einen werden nur 95% entfernt, der Rest reicht, um die Krankheit erneut ausbrechen zu lassen. Zum anderen wird für den Transport der CRISPR-DNS ein Virus verwendet, der im Körper eine Immunantwort auslöst und somit nur einmal eingesetzt werden kann. Außerdem stehen die Transportviren in Verdacht, Tumore auszulösen.14 Eine weitere Arbeitsgruppe hat dieses Jahr mittels CRISPR/Cas die regulatorischen Gene von HIV zerstört, sodass dieser nicht weiter in der Lage war, sich in den infizierten Zellen zu vermehren. Dafür haben sie einen Vektor konstruiert, der sowohl Cas9 als auch eine von sechs verschiedenen gRNSs codiert. Diese Vektoren wurden in Zellen transfiziert, die die regulatorischen Proteine des Virus (Tat und Rev) exprimieren. Dadurch wurde eine signifikante Erniedrigung in der Expression und Funktion der Proteine verzeichnet, ohne dass dies das Überleben der Zellen beeinträchtigt hat.15 Durch diese beiden Veröffentlichungen zeigt sich, dass es durch CRISPR eine Heilung für HIV-infizierte Menschen geben könnte.  Eine Nachricht, die viele Menschen geschockt hat, kam letzten Monat aus China. Ein Wissenschaftler behauptet, dass die zwei ersten, durch CRISPR/Cas veränderten Babys zur Welt gekommen sind. He Jiankui hat eine Frau mit Embryonen befruchtet, bei denen als Zygote mittels CRISPR/Cas das Gen CCR5 zerstört wurde. Dieses codiert ein Rezeptorprotein, an welches sich der HI-Virus bei Infektion anheftet und somit essentiell für diesen ist. Ziel dieser Arbeit war nicht, dass die Übertragung des Virus durch die Eltern verhindert wird, sondern vielmehr, dass die Kinder nicht das gleiche Schicksal erleiden müssen wie ihre Eltern und somit gegen den Virus resistent sind.16 Kritik kam hierbei von allen Seiten. Ein wohl wichtiges Argument ist, dass es auch einige HIV-Stämme gibt, die für die Infektion ein anderes Rezeptorprotein (CXCR4) verwenden.16 Zudem kommt der ethische Aspekt. In Deutschland, USA und vielen anderen Ländern sich derartige Manipulationen verboten.17 Die durch Genome-Editing versursachte Veränderungen werden auch an nachfolgende Generationen weitergegeben, somit sind die Risiken, die damit einher gehen, bisher kaum abzuschätzen.17 Deshalb bleibt der Einsatz dieser „Wunderwaffe“ nach wie vor mit großer Vorsicht zu genießen. Ansprechpartner: Kristina Schraml (kristina.schraml@biovariance.com) Quellen
Eine Nachricht, die viele Menschen geschockt hat, kam letzten Monat aus China. Ein Wissenschaftler behauptet, dass die zwei ersten, durch CRISPR/Cas veränderten Babys zur Welt gekommen sind. He Jiankui hat eine Frau mit Embryonen befruchtet, bei denen als Zygote mittels CRISPR/Cas das Gen CCR5 zerstört wurde. Dieses codiert ein Rezeptorprotein, an welches sich der HI-Virus bei Infektion anheftet und somit essentiell für diesen ist. Ziel dieser Arbeit war nicht, dass die Übertragung des Virus durch die Eltern verhindert wird, sondern vielmehr, dass die Kinder nicht das gleiche Schicksal erleiden müssen wie ihre Eltern und somit gegen den Virus resistent sind.16 Kritik kam hierbei von allen Seiten. Ein wohl wichtiges Argument ist, dass es auch einige HIV-Stämme gibt, die für die Infektion ein anderes Rezeptorprotein (CXCR4) verwenden.16 Zudem kommt der ethische Aspekt. In Deutschland, USA und vielen anderen Ländern sich derartige Manipulationen verboten.17 Die durch Genome-Editing versursachte Veränderungen werden auch an nachfolgende Generationen weitergegeben, somit sind die Risiken, die damit einher gehen, bisher kaum abzuschätzen.17 Deshalb bleibt der Einsatz dieser „Wunderwaffe“ nach wie vor mit großer Vorsicht zu genießen. Ansprechpartner: Kristina Schraml (kristina.schraml@biovariance.com) Quellen



